GeneDock HG Somatic 手册
Part 1 产品介绍
1. GeneDock HG Somatic 人全基因组体细胞突变标准化分析服务用途
全基因组测序 (Whole genome sequencing,WGS) 是指使用二代测序技术 (NGS) 对基因组的所有区域进行测序。相比于全外显子和捕获区域测序,全基因组可以获得更全面的信息,发现非编码区域的异常。成对样本 (matched samples) 可以鉴定出体细胞突变 (somatic mutations),常用于肿瘤研究领域。
GeneDock HG Somatic流程使用BWA、Sentieon软件TNseq,对全基因组成对测序样本数据,进行从fastq到vcf的分析(包含SNV和INDEL)。Sentieon TNseq是一款类似于GATK MuTect1/MuTect2的软件,包括TNsnv和TNhaplotyper,其中TNsnv与GATK MuTect1一致,可用于鉴定SNV,TNhaplotyper与GATK MuTect2一致,可用于鉴定SNV和INDEL。
GeneDock HG Somatic包括2个具体流程:
1)WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef:采用Sentieon软件TNsnv检测SNV(与MuTect1一致),Strelka软件检测INDEL。
2)WGS_Somatic_BWA-Sentieon2_ContainRef:采用Sentieon软件TNhaplotyper同时检测SNV和INDEL(与MuTect2一致)。
GeneDock的生信团队与数据计算团队共同努力,在数据传输优化、分析算法选择、计算资源选型等方面都进行了优化,并对分析关键步骤做了分布式处理,在保证同GATK MuTect1/MuTect2鉴定结果准确度一致的基础上,大大降低了用户的分析成本,缩短了运行时间。
Benchmark测试结果(http://biorxiv.org/content/biorxiv/early/2017/03/10/115717.full.pdf)显示,Sentieon软件TNseq在保证同GATK MuTect1/MuTect2软件鉴定结果准确度一致的基础上,比GATK MuTect1/MuTect2速度提升了约10倍(相同的硬件设备)。
更多信息:Sentieon官方网站:《 Sentieon TNseq, for consistent and confident somatic variant detection》
2. 整体步骤:
整体可分为3个部分:
1. 序列比对(Mapping):使用trimmomatic软件进行前处理,使用bwa mem进行比对,使用samtools对bam文件进行排序、格式转换等操作。
2. Bam文件前处理(Bam processing):使用Sentieon软件对重复序列进行处理(markdup/rmdup)和Base quality score recalibration(bqsr)。
3. SNV和INDEL检测(SNP and INDEL calling):1)WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef流程:使用Sentieon软件的TNsnv检测SNV变异,Strelka软件检测INDEL变异;2)WGS_Somatic_BWA-Sentieon2_ContainRef流程:使用Sentieon软件的TNhaplotyper对SNV和INDEL同时进行检测。
4. 对原始序列 (FASTQ) 和比对后序列 (BAM) 的基本计量参数进行统计:原始序列 (FASTQ)统计包括Reads数目、N所占比例、低质量碱基的比例、Q20、Q30、与Adaptor相关的reads比例,以及质量值和GC含量图等。比对后序列 (BAM)的统计包括:靶标区域所占比例、平均测序深度、重复区域比例、比对率、不同X数reads所占比例,以及目标区域测序深度图等,具体图表格式可以参考GeneDock 博客。
流程中步骤与GATK MuTect1的Best Practices或GATK MuTect2的Best Practices基本一致。
关于其中使用的knowsites输入文件也与GATK软件一致,可以参考这个链接。
【运行时间】
1)WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef流程:
2017年4月25日biolam_105测试80GB * 2 + 40GB * 2 的fastq.gz文件。深圳域:9小时。
2)WGS_Somatic_BWA-Sentieon2_ContainRef流程:
2017年3月11日genedock_wym测试80GB * 2 + 40GB * 2 的fastq.gz文件。北京域:8.5小时。
由于全基因组成对样本原始数据较大,因此给数据分析带来了很大的挑战。
本工作流利用“fastq按行拆分、bam按染色体分组拆分”的原理,可以大大缩短运行时间。
本流程中bam文件的16个染色体分组,具体情况如下(以hg19的染色体名为例):
- 第0组:chr1
- 第1组:chr2
- 第2组:chr3
- 第3组:chr4
- 第4组:chr5
- 第5组:chr6
- 第6组:chr7
- 第7组:chr8
- 第8组:chr9和chr10
- 第9组:chr11和chr12
- 第10组:chr13和chr14
- 第11组:chr15和chr16
- 第12组:chr17和chr18
- 第13组:chr19和chr20
- 第14组:chr21和chr22
- 第15组:chrX、chrY和chrM
- 同时输出unmapped bam文件
【准确性评估】
本流程步骤与GATK MuTect1/MuTect2的best practice基本一致,准确性评估请参考这个链接。
【注意事项】
本流程只适用于成对样本。
本流程需要是双端测序。
流程中会使用GATK官方推荐的reference、knowsites输入文件,下载自Broad的FTP。
如果输入fq文件是多个lane的,请注意read1和read2的顺序要一一对应。
关于此流程有任何问题,请您随时与我们的工程师联系。
Part 2 GeneDock HG Somatic操作教程
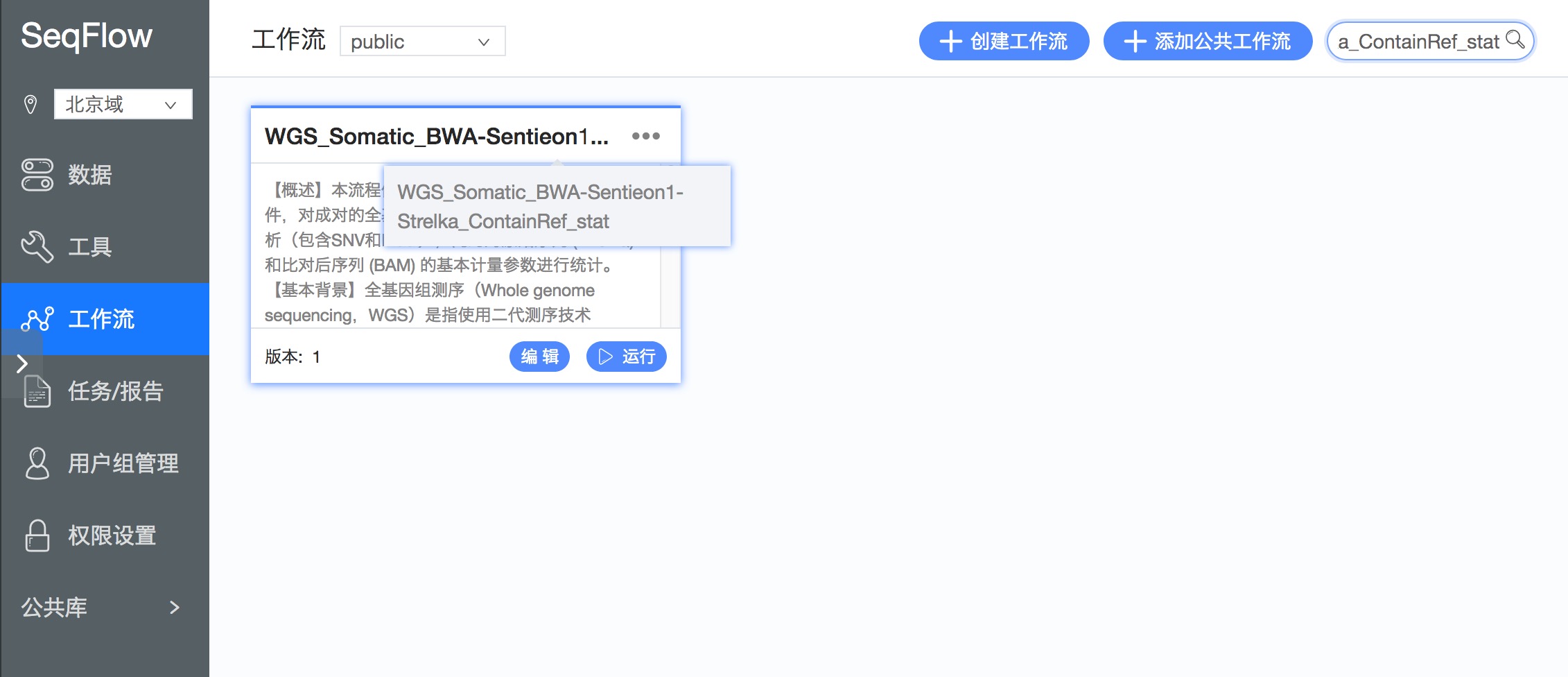
1.查看工作流详情:进入后台后,点击左侧工具栏中的工作流,在我的工作流标签中选择public,可以看到WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef_stat和WGS_Somatic_BWA-Sentieon2_ContainRef_stat工作流,点击“详情”,查看该工作流的介绍,包括:【概述】、【基本背景】、【整体步骤】、【运行时间】、【准确性评估】、【注意事项】等(如图1,图2);

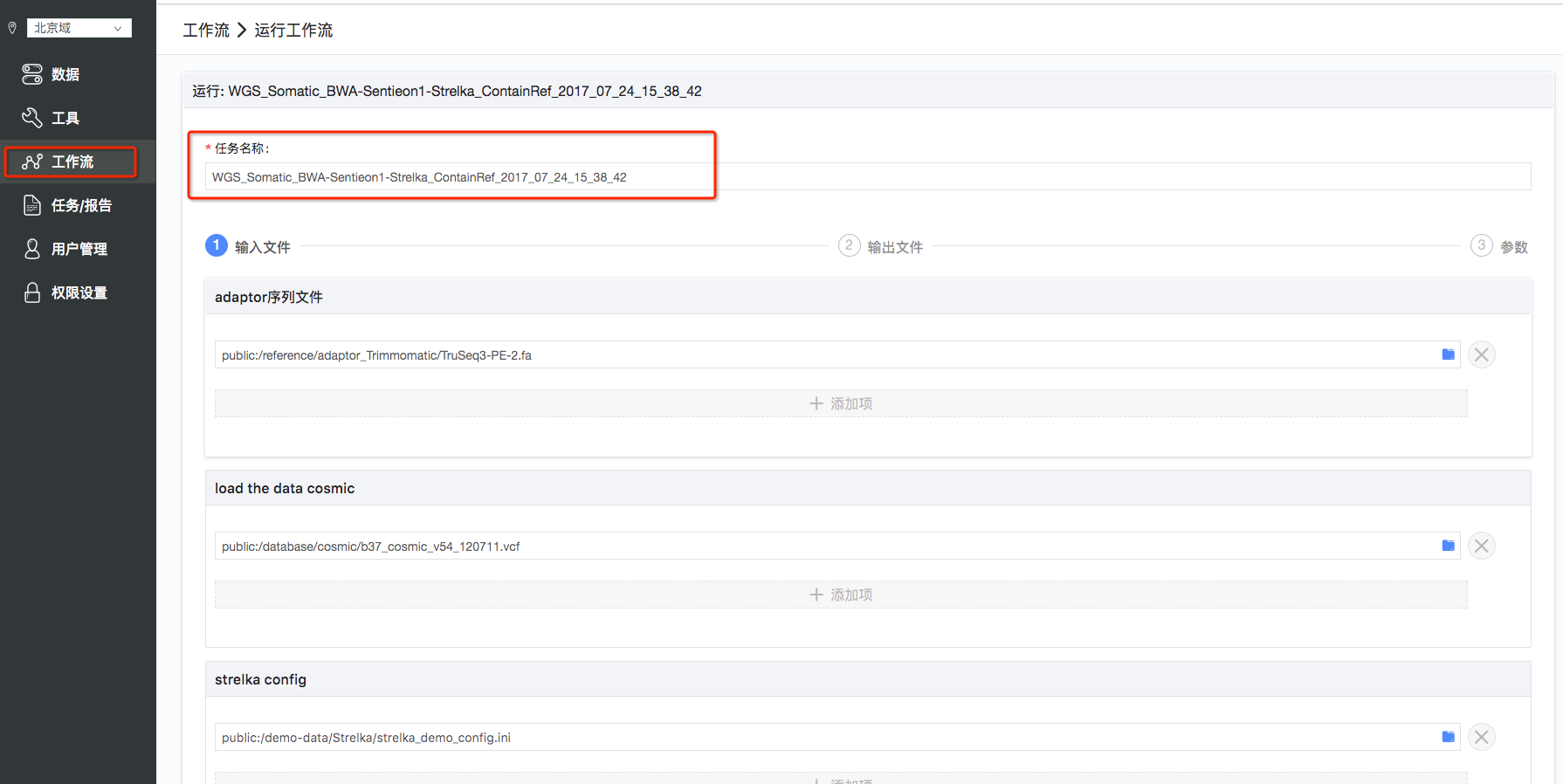
2.运行工作流:熟悉工作流的信息后,重复第一步骤,点击“运行”,进入到运行工作流界面(如图3),为了方便后续查找任务,可以修改“任务名称”;
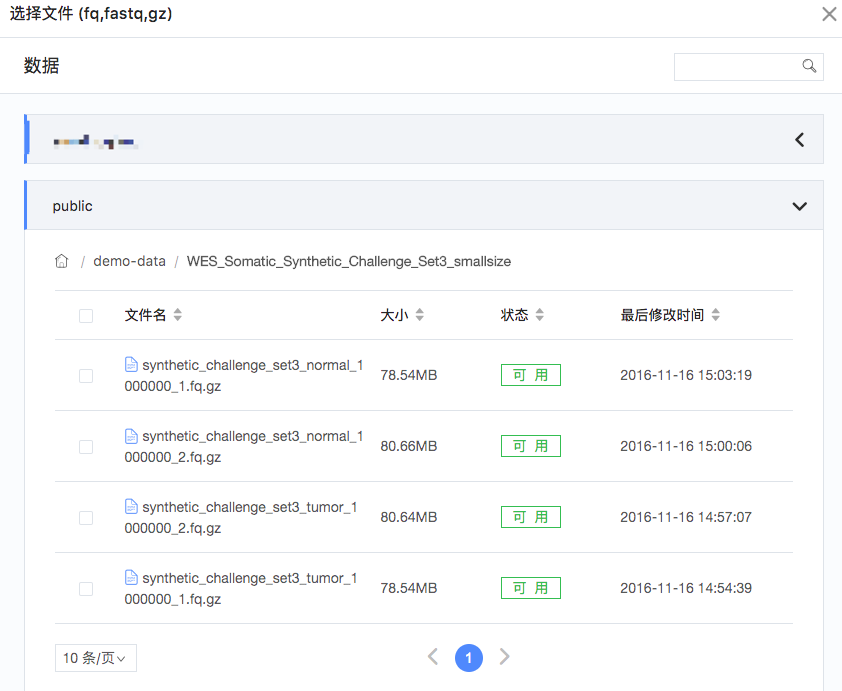
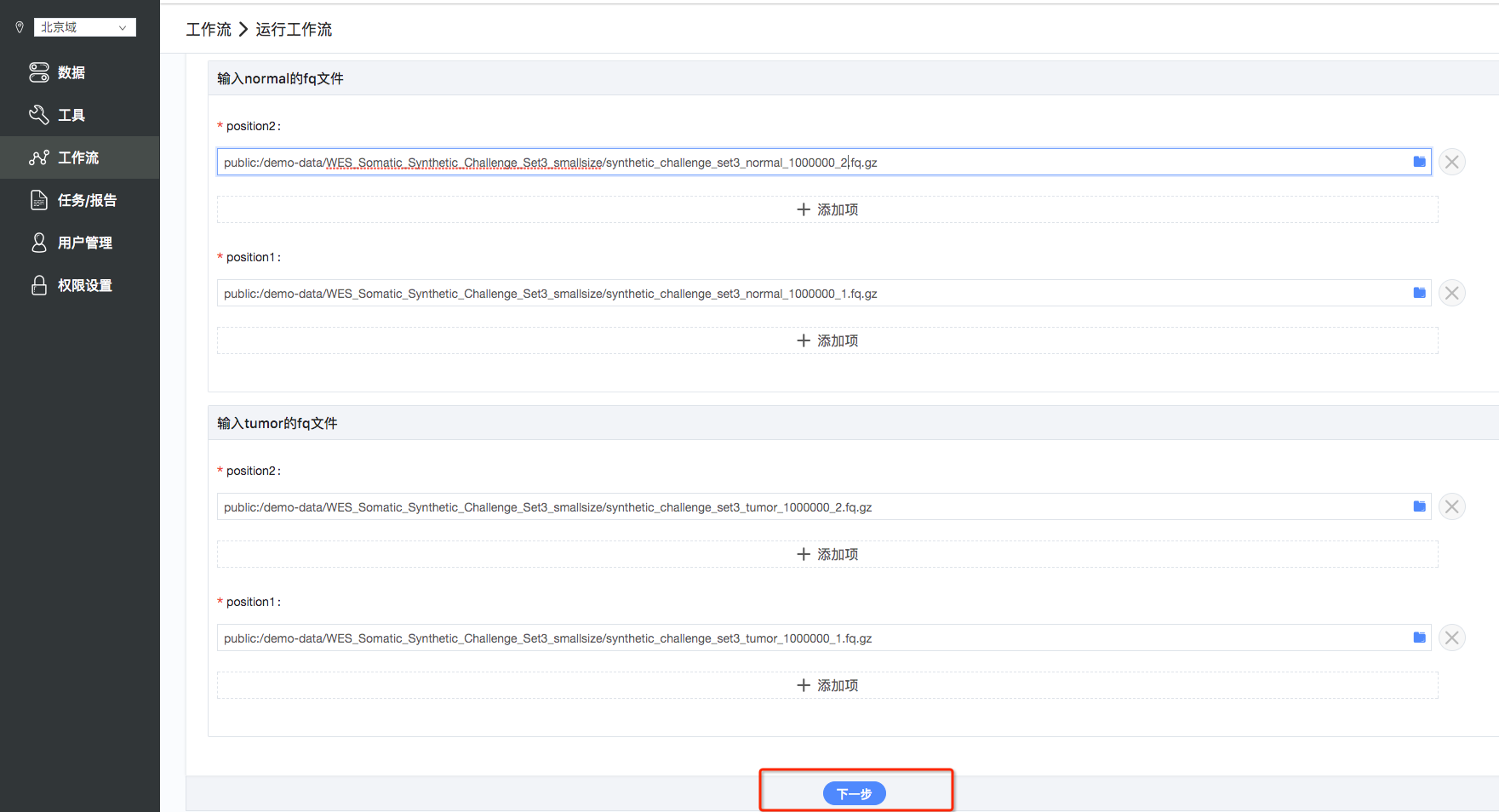
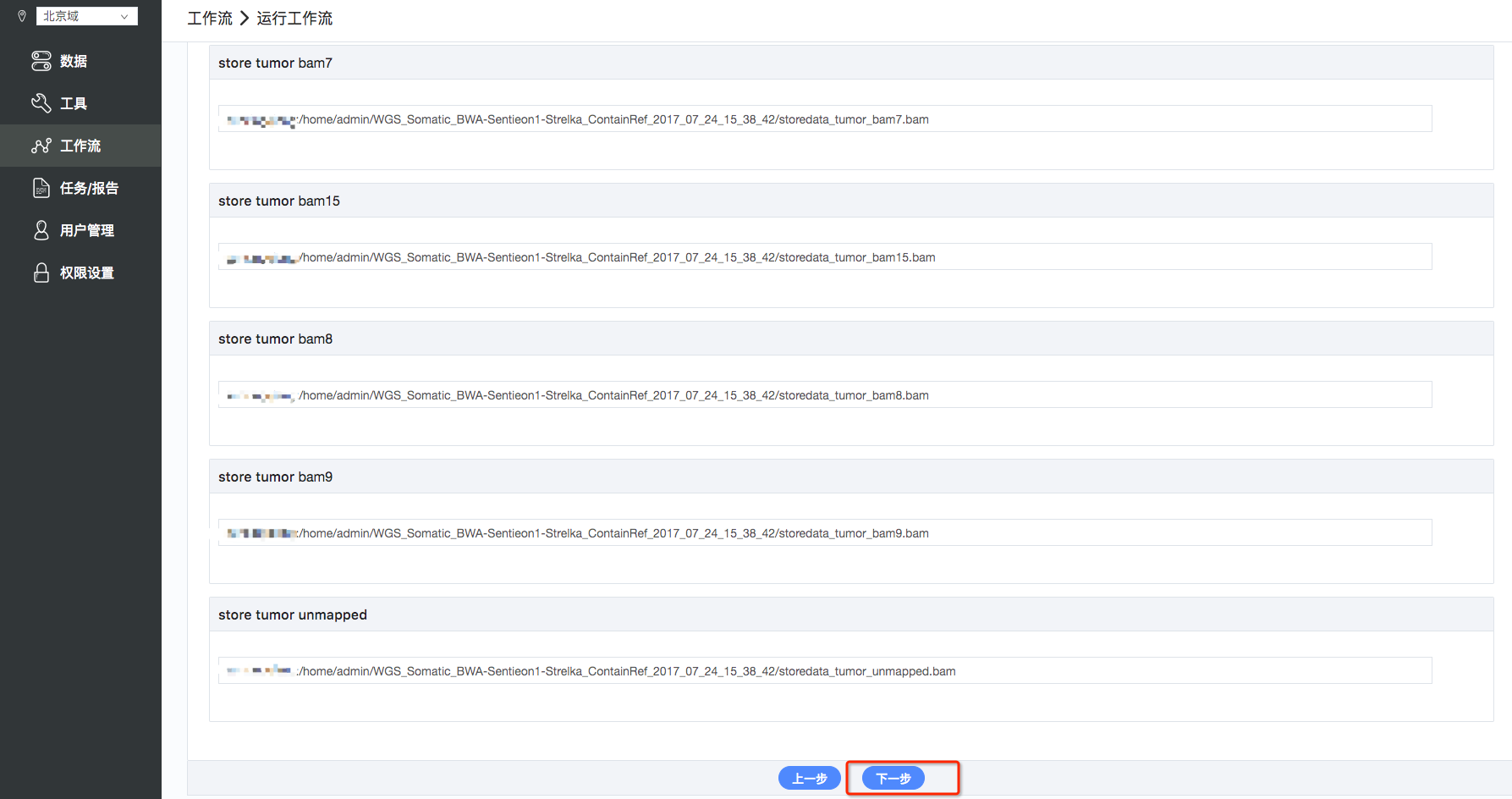
3.设置输入文件:需要输入1)Strelka config文件(仅:WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef流程);2)adaptor序列文件;3)tumor双端测序结果文件;4)normal双端测序结果文件;5)cosmic文件(默认b37参考文件)。点击输入文件条中对应的文件夹符号,可以看到对应的数据选择框,其中最上边可以看到对应的文件格式(如fq,fastq,gz),在您需要的文件前面操作栏选择“选取”,然后就可在输入文件条中看到选定的输入文件,所有输入文件都选择完毕后,点击“下一步”(如图4、图5);
4.设置输出文件:输出文件已经自动加载默认路径和文件名称,可以按照项目的目录情况自行修改, 如无需要可以不做修改,所有输出文件都选择完毕后,点击“下一步”(如图6);
5.设置参数:系统中会绑定默认参数,且会标明参数属于的工具名称,如有需要修改参数,将鼠标放置参数名称上可查看参数的说明,选择参数后可直接修改(如图7);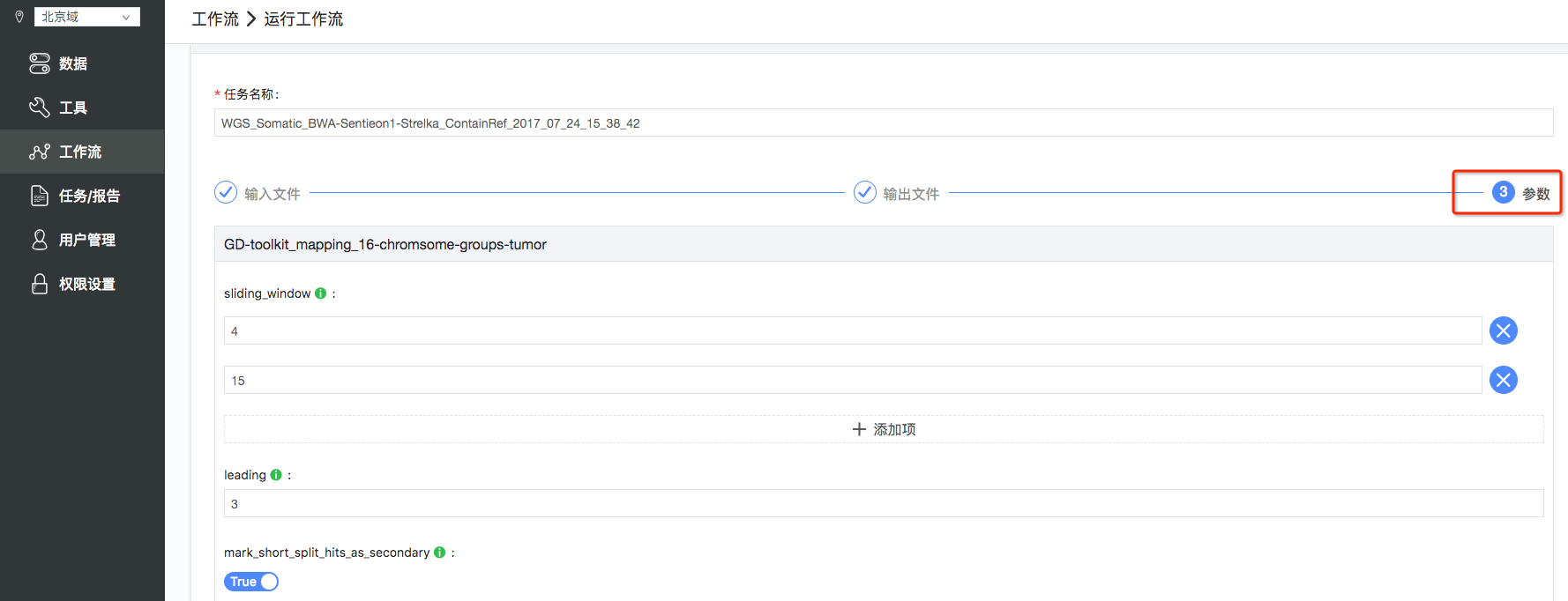
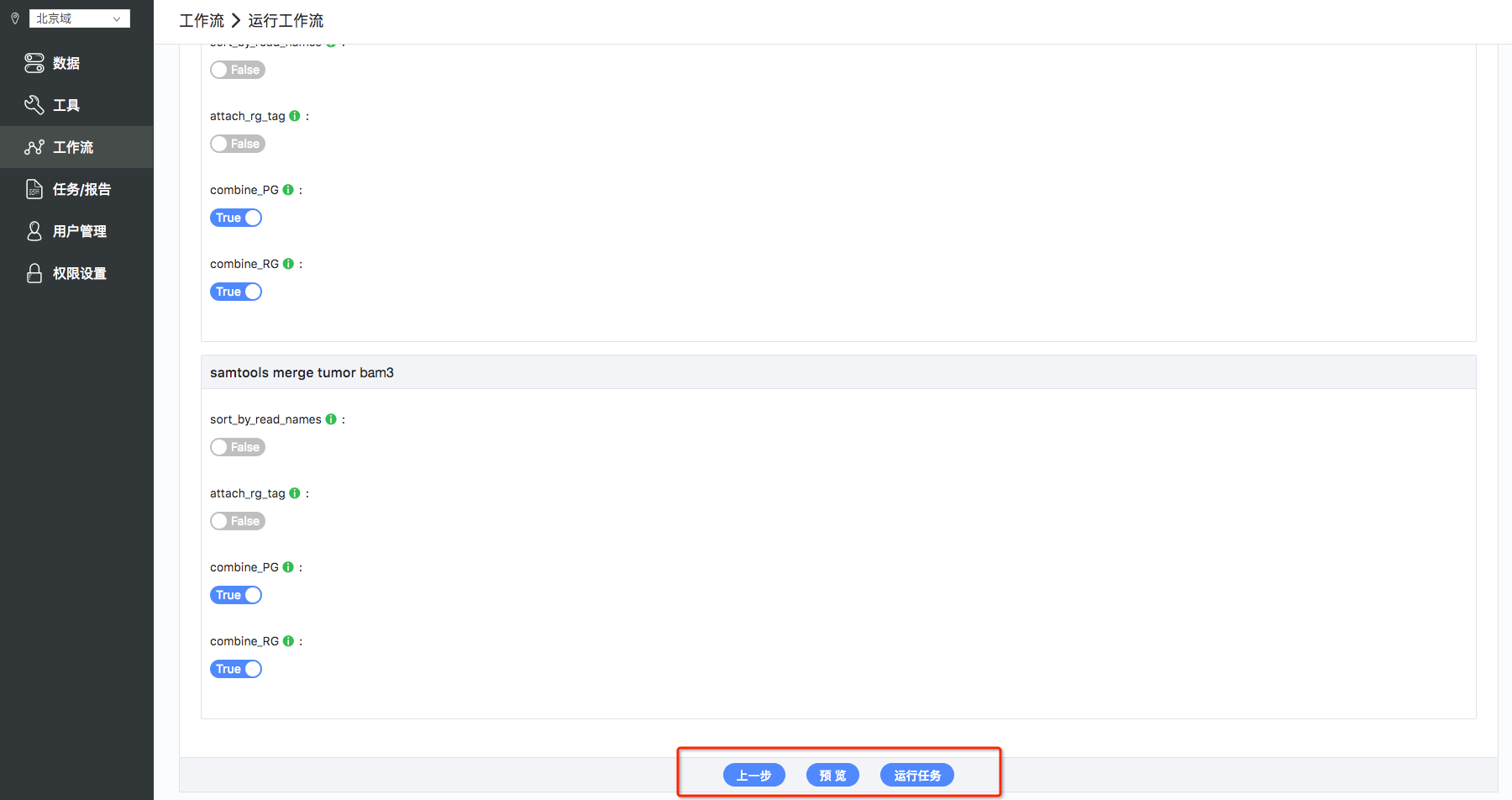
6.预览及运行工作流:填写好参数后,最后可以点击预览,确认前面的所有设置,确认好后点击‘运行任务’,开始运行工作流(如图8);
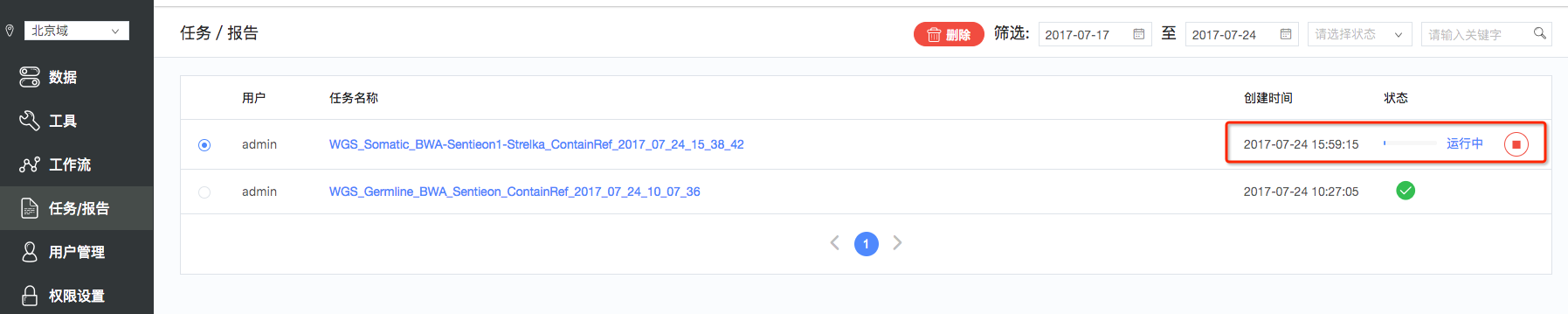
7.查看任务:几分钟后可以点击左侧工具栏中的“任务/报告”查看相应任务的运行状态。为了方面查看任务,用户可以按照任务创建和运行状态等筛选。另外,对于不需要的任务,可以删除该任务,(如图9)。
点击所要查询的任务,可以查看“参数”,“日志”,“报告”以及“相关文件下载和预览”。点击“参数“ ,可以看到输入,输出和相关参数,(如图10):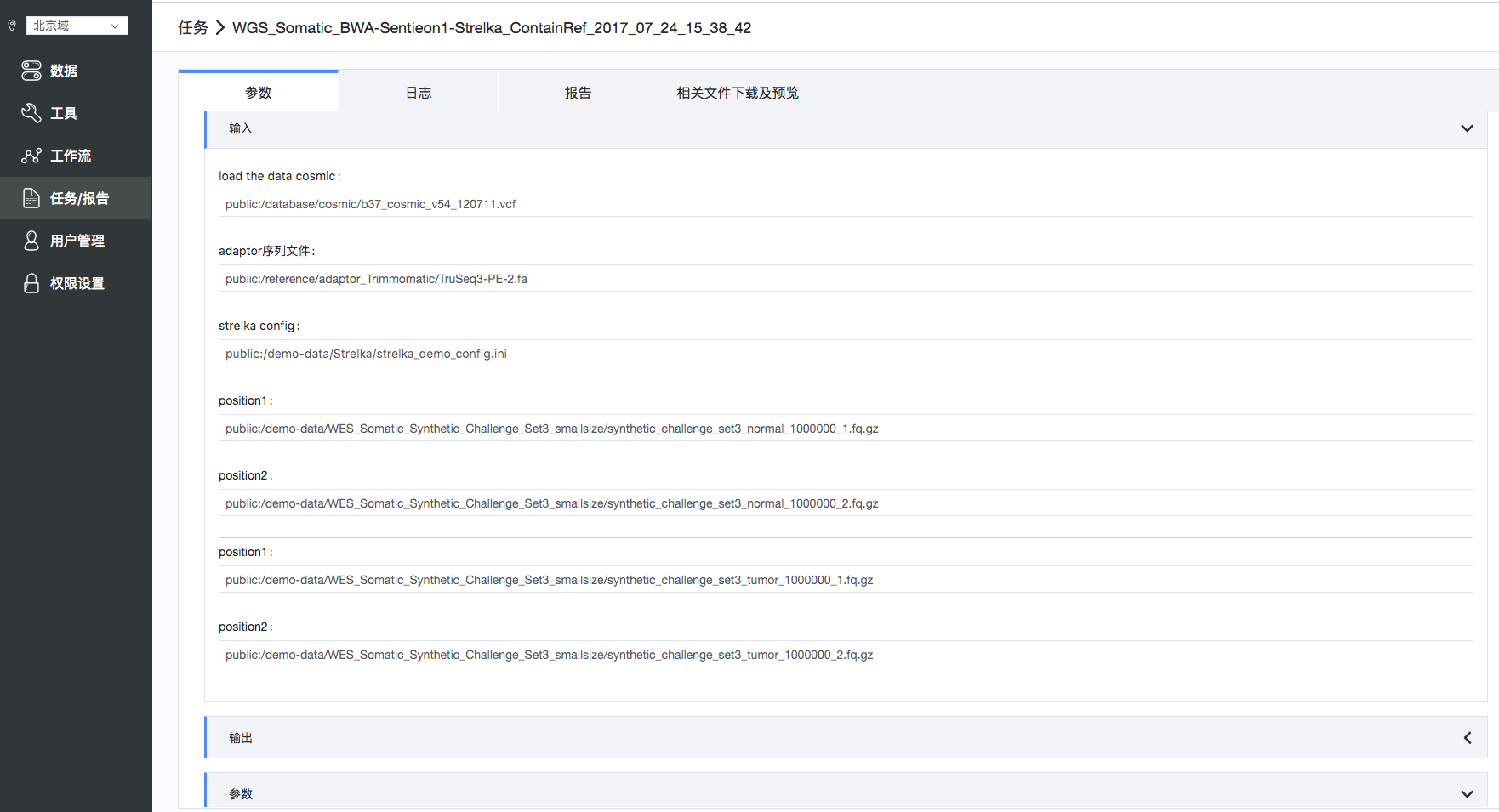
点击”日志“,可以看到每一步的完成状态。点击APP名称,可以看到部分输出日志,点击“下载日志”可以下载查看完整的输出日志(如图11);
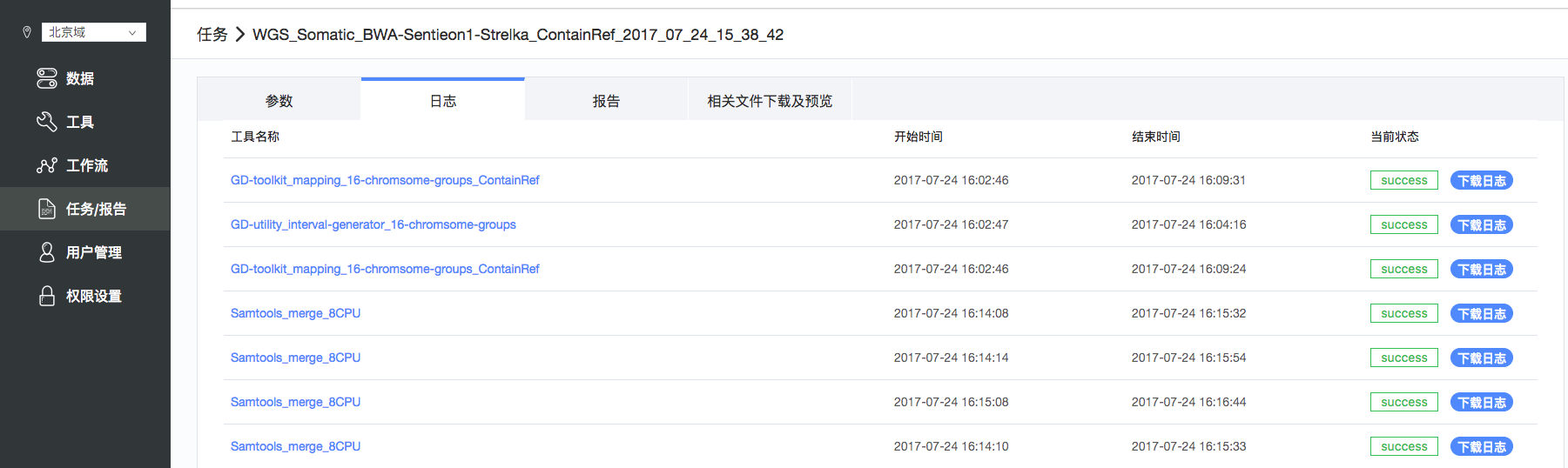
注:目前的工作流没有设置报告模板,因而报告不可查看,所以只能查看“相关文件下载和预览”。
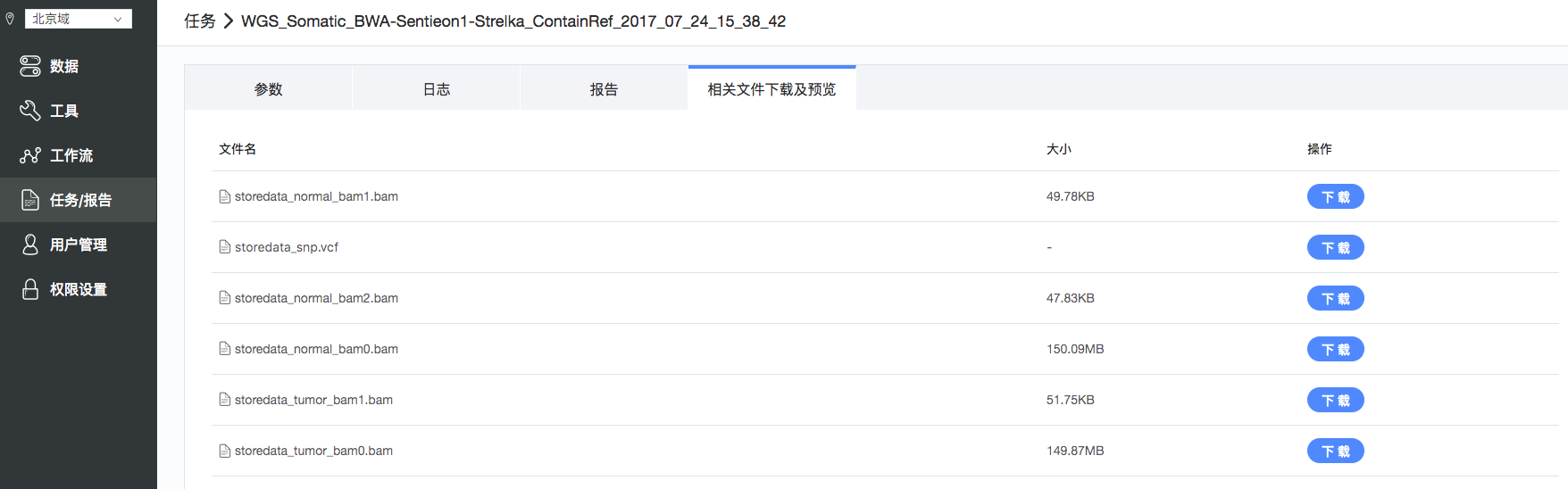
点击“相关文件下载和预览”,可以看到任务的结果文件。对于文本文件和png等图文件,支持在线预览和下载,超过100M的文件,建议按照下文使用客户端下载(如图12)。
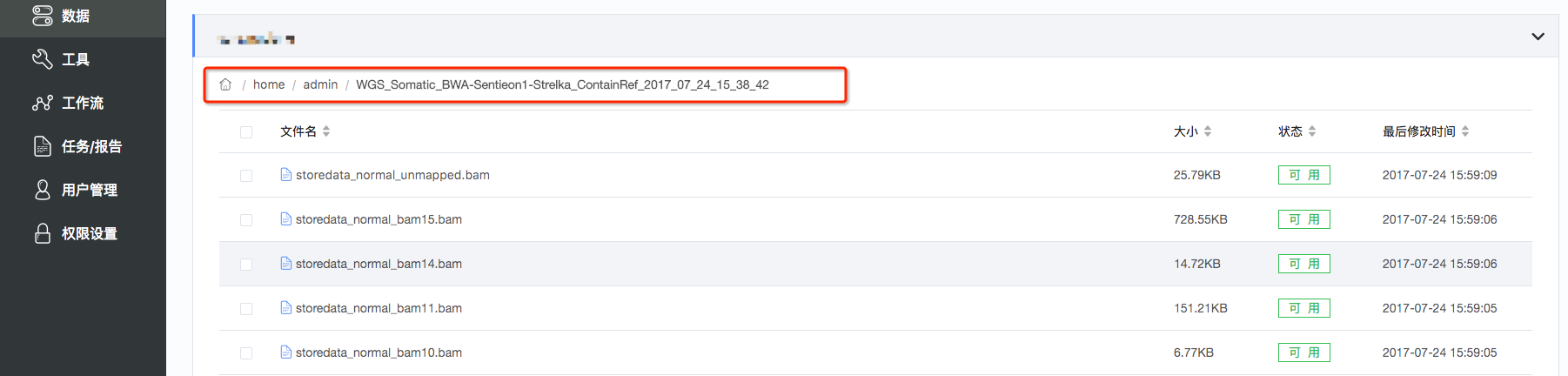
8.数据结果的查看和下载:点击左侧工具栏中的“数据”,选定对应的文件路径后可以看到结果文件,使用客户端进行下载(如图13)。
Part 3 参数解释
1. 工具:GD-toolkit_mapping_16-chromsome-groups-tumor/normal (共2个)
| 参数 | 软件 | 默认值 | 解释 |
|---|---|---|---|
| sliding_window | Trimmomatic | 4,15 | Windows的size是4个碱基,其平均碱基质量小于15,则切除 |
| leading | Trimmomatic | 3 | 切除首端碱基质量小于3的碱基或者N |
| mark_short_split_hits_as_secondary | Bwa-mem | Yes | 加入此参数用于将shorter split hits 标记为次优,有利于兼容 Picard、GATK |
| head_crop | Trimmomatic | 0 | 切除reads开始指定数目碱基 |
| crop | Trimmomatic | 10000 | 保留reads到指定的长度 |
| to_phred64 | Trimmomatic | False | 将碱基质量转换为pred64格式 |
| illumina_clip | Trimmomatic | 2,30,10 | 切除adapter序列:允许的最大mismatch数;palindrome模式下匹配碱基数阈值;simple模式下的匹配碱基数阈值 |
| to_phred33 | Trimmomatic | False | 将碱基质量转换为pred33格式 |
| read_group | Bwa-mem | sample | read group中sample名称,@RG\tID:{{parameters.read_group}}\tSM:{{parameters.read_group}}\tPL:illumina\tLB:lib |
| minlength | Trimmomatic | 36 | 最小的reads长度 |
| phred33 | Trimmomatic | False | 设置碱基的质量格式为phred33 |
| trailing | Trimmomatic | 3 | 切除尾端碱基质量小于指定值的碱基 |
| refname | Bwa-mem | b37 | 流程中使用的reference(可选hg19或者b37),reference文献下载自Broad的FTP |
2. 工具:samtools merge tumor/normal bam*(由于本流程分16份染色体且为成对样本,因此有2*16个bam,编号为0-15)
| 参数 | 软件 | 默认值 | 解释 |
|---|---|---|---|
| sort_by_read_names | samtools merge | False | 使输入的比对序列按照read的名字来排序,而不是染色体坐标 |
| attach_rg_tag | samtools merge | False | 给每个比对添加一个 RG 标签,标签的值是通过文件名来推测的 |
| combine_PG | samtools merge | True | 把 PG 标签和 colliding IDs 结合,而不是添加一个后缀去区分它们 |
| combine_RG | samtools merge | True | 把 RG 标签和 colliding IDs 结合,而不是添加一个后缀去区分它们 |
3. 工具:Sentieon_markdup_bqsr_ContainRef(共16个)
| 参数 | 软件 | 默认值 | 解释 |
|---|---|---|---|
| knowsites_BaseRecalibrator | sentieon | dbsnp_138.b37.vcf& Mills_and_1000G_gold_standard.indels.b37.vcf& 1000G_phase1.indels.b37.vcf |
Base Recalibrator步骤knowsites输入文件名称(可选hg19或者b37的相关文件,多个文件名称使用&连接)使用GATK官方推荐的knowsites输入文件,下载自Broad的FTP |
| rmdup | sentieon | False | 默认不去重复,只标注 |
| refname | sentieon | b37 | 流程中使用的reference(可选hg19或者b37),reference文献下载自Broad的FTP |
| thread | sentieon | 3 | 线程数(建议不要修改) |
4.1 工具:TNsnv(共16个)
仅WGS_Somatic_BWA-Sentieon1-Strelka_ContainRef流程
| 参数 | 软件 | 默认值 | 解释 |
|---|---|---|---|
| thread | sentieon TNsnv | 3 | 线程数(建议不要修改) |
| tumor_name | sentieon TNsnv | tumor | 肿瘤样本名称(需要和bwa的RG一致) |
| knownsites_IndelRealigner | sentieon TNsnv | Mills_and_1000G_gold_standard.indels.b37.vcf& 1000G_phase1.indels.b37.vcf |
knowsites输入文件名称(可选hg19或者b37的相关文件,多个文件名称使用&连接)使用GATK官方推荐的knowsites输入文件,下载自Broad的FTP |
| refname | sentieon TNsnv | b37 | 流程中使用的reference(可选hg19或者b37),reference文献下载自Broad的FTP |
| normal_name | sentieon TNsnv | normal | 正常样本名称(需要和bwa的RG一致) |
| dbsnp | sentieon TNsnv | dbsnp_138.b37.vcf | dbsnp文件名称(可选dbsnp_138.hg19.vcf或者dbsnp_138.b37.vcf),使用GATK官方推荐的knowsites输入文件,下载自Broad的FTP |
4.2 工具:Sentieon_TNhaplotyper_variantcalling_ContainRef(共16个)
仅WGS_Somatic_BWA-Sentieon2_ContainRef流程
| 参数 | 软件 | 默认值 | 解释 |
|---|---|---|---|
| thread | sentieon TNsnv | 3 | 线程数(建议不要修改) |
| tumor_name | sentieon TNsnv | tumor | 肿瘤样本名称(需要和bwa的RG一致) |
| knownsites_IndelRealigner | sentieon TNsnv | Mills_and_1000G_gold_standard.indels.b37.vcf& 1000G_phase1.indels.b37.vcf |
knowsites输入文件名称(可选hg19或者b37的相关文件,多个文件名称使用&连接)使用GATK官方推荐的knowsites输入文件,下载自Broad的FTP |
| refname | sentieon TNsnv | b37 | 流程中使用的reference(可选hg19或者b37),reference文献下载自Broad的FTP |
| normal_name | sentieon TNsnv | normal | 正常样本名称(需要和bwa的RG一致) |
| dbsnp | sentieon TNsnv | dbsnp_138.b37.vcf | dbsnp文件名称(可选dbsnp_138.hg19.vcf或者dbsnp_138.b37.vcf),使用GATK官方推荐的knowsites输入文件,下载自Broad的FTP |